Assoziation struktureller Variationen (CNVs)
Lange Zeit war es Konsens, dass Deletionssyndrome wie das auf Chromosom 22 die Ausnahme in der Psychiatrie darstellen.
Die 22q11.2 Mikrodeletion ist verantwortlich für das Velocardiofaciale Syndrom (VCFS), welches mit kognitiven Defiziten und Psychose einhergeht. Innerhalb der deletierten Region befinden sich u.a. das COMT (Catechol-O-Methyl transferase)- Gen, sowie TBX1 und GNBL1, für die eine Beteiligung an der Hirnfunktion und –entwicklung beschrieben wurde. Trotz zahlreicher Studien ist aber nach wie vor unklar, welches der deletierten Gene für die Prädisposition für die Erkrankung verantwortlich ist.
Die balancierte Translokation 22(1;11)(q42.1;q14.3) führt zur Teilung des DISC1 (Disrupted in Schizophrenia 1)-Gens, welches bereits 1990 mit psychiatrischen Erkrankungen in Zusammenhang gebracht wurde (St Clair et al. 1990). Es beeinflusst die Zellproliferation, Migration, Dendritogenese, Synaptogenese und die intrazelluläre Signaltransduktion (Randall et al. 2014; Porteous et al. 2014).
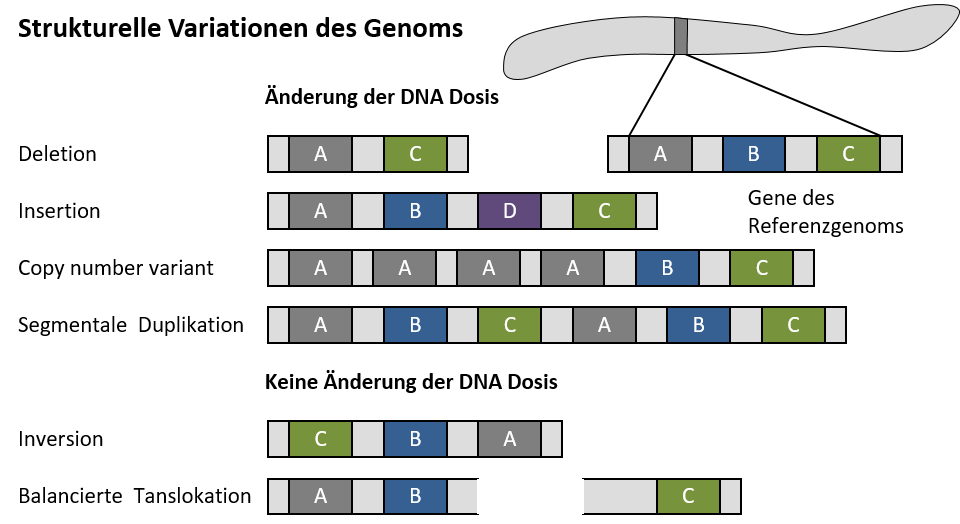
Mittlerweise hat sich dieses Bild geändert. Durch den Einsatz neuer Genotypisierungs- und Sequenziertechnologien, welche Lücken in der Auflösung der cytogenetischen Genomkarte (> 2 Mb) zur Sequenz-basierten Genomkarte bis auf wenige Ausnahmen geschlossen haben, ist es möglich strukturelle Variationen in allen Größenbereichen verlässlich zu identifizieren. Das betrifft u.a. auch CNVs (Copy Number Variations, >1kb), Duplikationen oder Deletionen (Grayton et al. 2012).
Mit diesem Ziel untersuchten Stefansson, Rujescu und Cichon et al. (2008) als eine der ersten eine Stichprobe aus insgesamt über 50.000 Individuen und ermittelten dabei 3 chromosomale Regionen, die mit der Schizophrenie hinsichtlich CNVs assoziiert waren (Deletionen auf 1q21.1; 15q11.2; 15q13.3).
Bemerkenswert hierbei ist, dass diese Varianten zwar nur von wenigen Personen getragen werden, dafür aber das Risiko von Werten mit bis zu 15 bedeutend höher liegt als z.B. bei SNP-Ergebnissen.
Interessanterweise konnten alle 3 Befunde repliziert werden. Zwei hiervon in einer groß angelegten Studie des International Schizophrenia Consortiums (ISC 2008) und eine weitere in der Arbeit von Kirov et al. (2009).

Stefansson H*, Rujescu D*, Cichon S* (*contributed equally) et al. (2008) Large recurrent microdeletions associated with schizophrenia. Nature. Sep 11; 455(7210): 232-6.
Neben den Deletionen auf den Chromosomen 1q21.1; 15q11.2 und 15q13.3 konnten auf diese Weise auch andere Regionen ermittelt werden.
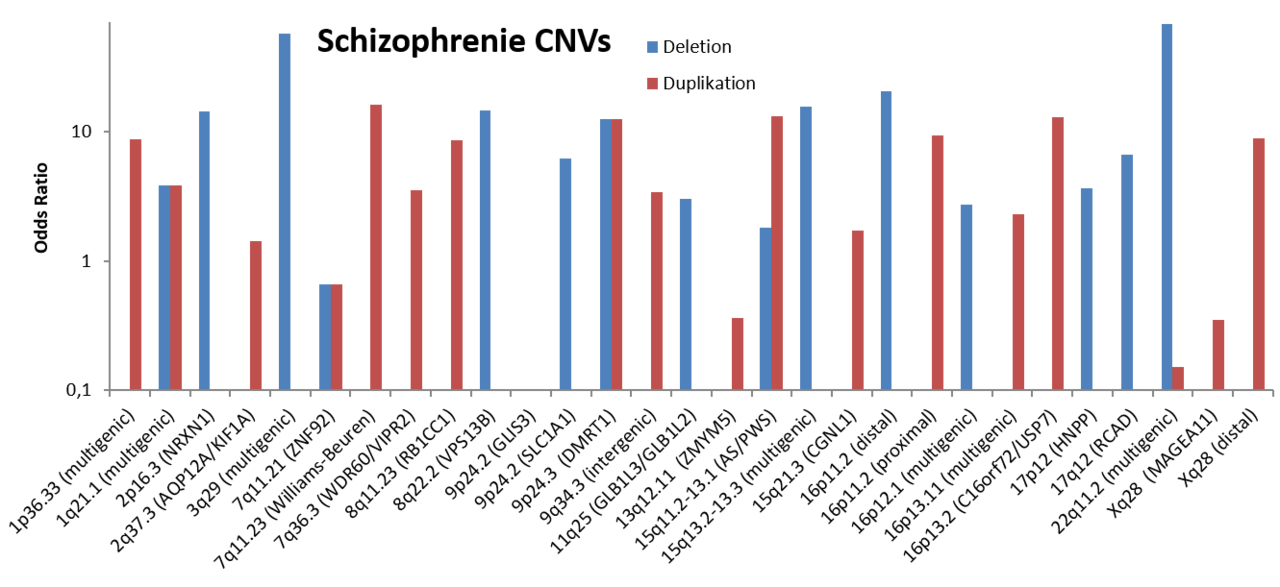
Daneben ergaben sich Hinweise für CNVs auf den Chromosomen 3q29, 16p11.2, 17p12 und 17q12 mit dem stärksten Effekt für 3q29 (OR= 57,65, CI 7,58-438,44, p=1,5x10-9), die in einer Metaanalyse von Rees et al. (2014) repliziert werden konnten.
Für das Neurexin 1 Gen (NRXN1, 2p16.3) gab es erste Hinweise aus einer Assoziation von Deletionen mit Autismus, sowie in 2 Familien mit Schizophrenie (Kirov et al. 2008, Walsh et al. 2008).
In einer Studie des SGENE+ Konsortiums mit 2.977 Schizophreniepatienten und 33.746 Kontrollen wurde durch die signifikante Anreicherung von CNVs in Patienten festgestellt, dass es zu r Unterbrechung in Exons kam, ein weiterer Nachweis für die Bedeutung des Neurexin 1 Gens in der Schizophrenie (Rujescu et al. 2009).

Rujescu D et al. (2009) Disruption of the neurexin 1 gene is associated with schizophrenia. Hum Mol Genet. 2009 Mar 1; 18(5): 988-96.
Des Weiteren konnte die Bedeutung großer Exons des NRXN1 Gens unterbrechender CNVs (> 100kb) durch eine Anreicherung in Patienten in einer Metaanalyse verdeutlicht werden (Kirov et al. 2009). Bestätigt wurden diese Ergebnisse auch in einer weiteren Studie (Levinson et al. 2011) und einer Metaanalyse (Rees et al. 2014).

Kirov G et al. (2009) Neurexin 1 (NRXN1) deletions in schizophrenia. Schizophr Bull. 2009 Sep; 35(5): 851-4.
Eine Anreicherung von CNVs bei Schizophreniepatienten veranlasste Kirov et al. Eltern- Kind- Trios auf de novo CNVs zu screenen. Auch in dieser Stichprobe konnte eine signifikante Häufung von de novo CNVs in bereits bekannten Regionen identifiziert werden (3q29, 15q11.2, 15q13.3, 16p11.2) (Kirov et al. 2012). Insgesamt war die CNV- Last in den Patienten signifikant höher als in den Kontrollen. Nominal signifikante Deletionen konnten u.a. in den Genen DLG2 (p=0,02), MSRA (p=0,03) und EHMT1 (p=0,055) detektiert werden.

Marshall CR et al. (2017) Contribution of copy number variants to schizophrenia from a genome-wide study of 41,321 subjects. Nat Genet. Jan:49(1):27-35.
In einer weiteren Studie mit 21.094 Patienten und 20.227 Kontrollen konnte diese Häufung von CNVs in Patienten erneut bestätigt werden. Des Weiteren waren 8 bereits bekannte Loci genomweit signifikant (Deletionen bei 1q21.1, 2p16.3 (NRXN1), 3q29, 15q13.3, 16p11.2 (distal), 22q11.2 und Duplikationen bei 7q11.23 und 16p11.2 (proximal)). Für weitere bekannte und neue Loci (Duplikationen bei 13q12.11, Xq28 und Xq28 distal, 7q36.3, Deletion bei 8q22.2, sowohl Duplikationen als auch Deletionen bei 7q11.21 und 9p24.3) konnten Hinweise gefunden werden. Eine genauere Analyse der Regionen ergab eine Anreicherung speziell von solchen Genen, die mit synaptischer Funktion und neurobehavioralen Phänotypen in Mausmodellen in Zusammenhang stehen (Marshall et al. 2017).
Insgesamt gibt es sowohl für Deletionen als auch für Duplikation Hinweise auf eine Beteiligung an der Pathophysiologie der Schizophrenie.
Die Häufung von CNVs in Patienten mit Psychose gibt Anlass zur Hoffnung, dass diese Deletionen und Duplikationen direkt zur Pathophysiologie und damit einer möglichen Therapie hinführen werden.
Dies wird unter anderem dadurch bestärkt, dass CNVs an der Pathophysiologie weiterer Erkrankungen wie beispielsweise Mentaler Retardierung oder Autismus beteiligt zu sein scheinen (Grayton et al. 2012). Epigenetische Einflüsse scheinen hier teilweise eine diskriminierende Rolle zwischen den Erkrankungen zu spielen (Isles et al. 2016).